L'objectif de ce TP est créer une simple base de données de vecteurs, comme décrit dans notre article "Sous le capot des bases de données vectorielles".
Bien évidemment, le but ici n'est pas de créer une base de données performante utilisable en production, mais plutôt de l'implémenter pas à pas pour en décortiquer chaque brique.
N'hésitez pas à vous référer à l'article pour mieux comprendre les concepts implémentés, ou à regarder le repository complet pour voir l'implémentation finale.
A noter que des tests unitaires ont été implémentés dans le repository, mais nous n'allons pas les recoder dans le TP. Libre à vous d'aller les voir sur la branche main et de les ajouter à votre implémentation ;-).
Pour référence, l'arborescence finale doit ressembler à ceci :
.
└── simple_vector_db
├── simple_vector_db
| ├── quantization
| | ├── __init__.py
| | ├── vector_quantizer.py
| ├── __init__.py
| ├── distances.py
| ├── numpy_array_adapter.py
| ├── vector_db_in_memory.py
| ├── vector_db_sqlite.py
| ├── vector_db_sqlite_pq.py
├── utils
| ├── flex_logging.py
main_in_memory.py
main_quantizer.py
main_sqlite.py
main_sqlite_pq.py
poetry.lock
pyproject.toml
Dans un premier temps, nous allons installer le dépendances nécessaires à notre projet. Pour cela, installez scikit-learn, numpy, sqlalchemy, matplotlib, dataget dans votre environnement Python. Il y a plusieurs façons de le faire : Conda, Pipenv, Poetry, etc. Libre à vous d'utiliser la méthode que vous préférez. Ici, nous allons utiliser Poetry en créant un fichier pyproject.toml et en y ajoutant les dépendances nécessaires :
# pyproject.toml
[tool.poetry]
name = "simple-vector-db"
version = "0.1.0"
description = ""
authors = ["Aurelien Massiot <aurelien.massiot@octo.com>", "Philippe Stepniewski <philippe.stepniewski@octo.com>"]
readme = "README.md"
packages = [{include = "simple_vector_db"}]
[tool.poetry.dependencies]
python = "^3.9"
scikit-learn = "^1.3.0"
sqlalchemy = "^2.0.20"
matplotlib = "^3.8.0"
dataget = "^0.4.15"
[tool.poetry.group.dev.dependencies]
pytest = "^7.4.1"
black = "^23.7.0"
[build-system]
requires = ["poetry-core"]
build-backend = "poetry.core.masonry.api"
Ensuite, un poetry install devrait installer les dépendances nécessaires dans votre environnement Python, poetry shell devrait vous permettre d'activer votre environnement.
Pour calculer des distances entre les vecteurs, il existe plusieurs méthodes et nous allons en implémenter deux : la distance euclidienne et la similarité cosinus. Pour cela, commençons par créer un fichier distances.py.
Les méthodes cosine_similarity et euclidean_distance prennent toutes les deux en entrée deux vecteurs et retournent un nombre entre 0 et 1. Plus ce nombre est proche de 1, plus les vecteurs sont similaires.
# simple_vector_db/distances.py
import numpy as np
def cosine_similarity(v1: np.ndarray, v2: np.ndarray) -> float:
dot_product = np.dot(v1, v2)
norm_v1 = np.linalg.norm(v1)
norm_v2 = np.linalg.norm(v2)
return dot_product / (norm_v1 * norm_v2)
def euclidean_distance(v1: np.ndarray, v2: np.ndarray) -> float:
return np.linalg.norm(v1 - v2)
Maintenant que nous avons des méthodes pour calculer des distances entre vecteurs, nous allons implémenter une base de données vecteurs en mémoire. Pour cela, nous allons créer un fichier simple_vector_db/vector_db_in_memory.py et y créer une classe InMemoryVectorDB.
Ici, nous implémentons les méthodes insert, search et retrieve en utilisant un dictionnaire python pour stocker les vecteurs. La clé de chaque vecteur est son index dans le dictionnaire.
Pour chercher les vecteurs les plus proches d'une requête, nous calculons la distance entre la requête et tous les vecteurs de la base de données, puis nous trions les résultats par ordre décroissant de distance et nous ne gardons que les k premiers. Il est donc possible ici d'utiliser la distance eucliennne ou la similarité cosinus.
#simple_vector_db/vector_db_in_memory.py
from collections import defaultdict
from typing import List, Tuple
import numpy as np
from simple_vector_db.distances import cosine_similarity
class VectorDBInMemory():
def __init__(self):
self.vectors = defaultdict(np.ndarray)
def insert(self, vectors: list[np.ndarray]) -> None:
for i in range(len(vectors)):
self.vectors[i] = vectors[i]
def search(self, query_vector: np.ndarray, k: int) -> List[Tuple[str, float]]:
similarities = [
(key, cosine_similarity(query_vector, vector))
for key, vector in self.vectors.items()
]
# similarities = [(key, euclidean_distance(query_vector, vector)) for key, vector in self.vectors.items()]
similarities.sort(key=lambda x: x[1], reverse=True)
return similarities[:k]
def retrieve(self, key: int) -> np.ndarray:
return self.vectors.get(key, None)
Pour pouvoir suivre l'exécution de notre code, nous allons ajouter un peu de logging coloré (copyright à notre cher Octo Thomas) . Pour cela, nous allons créer un fichier utils/flex_logging.py et y créer un handler qui permettra d'afficher les logs dans la console. C'est clairement un nice-to-have, mais autant faire les choses bien :
# utils/flex_logging.py
import logging
import sys
class CustomFormatter(logging.Formatter):
green = "\x1b[32m"
yellow = "\x1b[33m"
red = "\x1b[31;20m"
reset = "\x1b[0m"
format = "%(asctime)s - %(levelname)s - %(message)s - (%(filename)s:%(lineno)s)"
FORMATS = {
logging.INFO: green + format + reset,
logging.WARNING: yellow + format + reset,
logging.ERROR: red + format + reset,
}
def format(self, record):
log_fmt = self.FORMATS.get(record.levelno)
formatter = logging.Formatter(log_fmt)
return formatter.format(record)
stream_handler = logging.StreamHandler(sys.stdout)
stream_handler.setLevel(logging.INFO)
stream_handler.setFormatter(CustomFormatter())
Maintenant que nous avons implémenté notre première base de données vecteurs, testons la. Nous allons créer un fichier main_in_memory.py dans lequel nous allons pouvoir tester notre implémentation.
Pour cela, nous allons créer une base de données vecteurs en mémoire, y insérer 3 vecteurs sous forme de numpy array, en retrouver un par sa clé et chercher les k vecteurs les plus similaires de notre vecteur requête.
N'hésitez pas à changer les valeurs des vecteurs (les vecteurs insérés vectors_to_insert au préalable et le vecteur de requête QUERY_VECTOR).
Notez l'import de notre librairie de logging et l'utilisation de notre handler stream_handler pour afficher les logs dans la console.
A ce stade, l'arborescence de notre projet devrait ressembler à cela :
.
└── simple_vector_db
├── simple_vector_db
| ├── __init__.py
| ├── distances.py
| ├── vector_db_in_memory.py
├── utils
| ├── flex_logging.py
poetry.lock
pyproject.toml
main_in_memory.py
# main_in_memory.py
import logging
import numpy as np
from simple_vector_db.vector_db_in_memory import VectorDBInMemory
from utils.flex_logging import stream_handler
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
vector_db = VectorDBInMemory()
QUERY_VECTOR = np.array([0.1, 0.2, 0.3])
K_SIMILAR_VECTORS = 3
def perform_search_in_memory():
vectors_to_insert = [
np.array([10, 20, 30]),
np.array([-1, -2, -3]),
np.array([0.3, 0.3, 0.3]),
]
vector_db.insert(vectors_to_insert)
retrieved_vector = vector_db.retrieve(1)
logger.info(f"Retrieved vectors: {retrieved_vector}")
similar_vectors = vector_db.search(query_vector=QUERY_VECTOR, k=K_SIMILAR_VECTORS)
logger.info(f"Most {K_SIMILAR_VECTORS} Similar vectors: {similar_vectors}")
if __name__ == "__main__":
perform_search_in_memory()
Et tadaaa !
Nous pouvons voir que les k vecteurs les plus similaires ont été retournés, ordonnés par ordre décroissant de similarité.

Nous allons désormais passer aux choses sérieuses et implémenter une base de données vecteurs persistante.
Avant d'implémenter notre base de données persistante, nous allons d'abord créer un petit utilitaire nous permettant de mesurer le temps d'exécution de nos méthodes. Pour cela, nous allons créer un fichier utils/timer.py et y créer un décorateur timer qui prend en entrée une fonction et retourne une fonction qui mesure le temps d'exécution de la fonction passée en entrée.
Créez un fichier utils/timing.py et implémentez le décorateur timer :
# utils/timing.py
import logging
import time
from functools import wraps
from utils.flex_logging import stream_handler
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
def timeit(func):
@wraps(func)
def timeit_wrapper(*args, **kwargs):
start_time = time.perf_counter()
result = func(*args, **kwargs)
end_time = time.perf_counter()
total_time = end_time - start_time
logger.info(
f"Function {func.__name__}{args} {kwargs} Took {total_time:.4f} seconds"
)
return result
return timeit_wrapper
Nous allons maintenant créer une base de données SQLite avec SQLAlchemy. SQLAlchemy va grandement nous faciliter la tâche en tant qu'ORM. Pour cela, nous allons créer un fichier simple_vector_db/vector_db_sqlite.py et y créer une classe VectorDBSQLite.
L'initialisation de cet objet va nous permettre d'instancier la connexion à la base de données SQLite :
# simple_vector_db/vector_db_sqlite.py
import sqlite3
from sqlalchemy import create_engine
from sqlalchemy.orm import declarative_base
from sqlalchemy.orm import sessionmaker
from simple_vector_db.distances import cosine_similarity, euclidean_distance
Base = declarative_base()
class VectorDBSQLite():
def __init__(self, db_filename: str):
self.engine = create_engine(f"sqlite:///{db_filename}", connect_args={'detect_types': sqlite3.PARSE_DECLTYPES})
Base.metadata.create_all(self.engine)
Session = sessionmaker(bind=self.engine)
self.session = Session()
self.metric = {"metric": cosine_similarity, "reverse_sort": True}
def set_metric(self, metric_name: str = "cosine"):
if metric_name == "cosine":
self.metric["metric"] = cosine_similarity
self.metric["reverse_sort"] = True
elif metric_name == "euclidean":
self.metric["metric"] = euclidean_distance
self.metric["reverse_sort"] = False
Dans ce même fichier, nous allons ajouter une classe Vector qui va nous permettre de représenter un vecteur dans notre base de données. Cette classe va hériter de la classe Base de SQLAlchemy et va contenir un attribut id qui sera la clé primaire de notre table et un attribut vector qui sera le vecteur stocké dans la base de données.
from sqlalchemy import Column, Integer
from simple_vector_db.numpy_array_adapter import NumpyArrayAdapter
class Vector(Base):
__tablename__ = "vectors"
id = Column(Integer, primary_key=True)
data = Column(NumpyArrayAdapter)
def __init__(self, data):
self.data = data
Nous pouvons voir qu'ici, notre vecteur est de type NumpyArrayAdapter, et qu'il est importé depuis simple_vector_db.numpy_array_adapter. Ce fichier n'existe pas ? C'est normal, nous allons le créer tout de suite.
Il n'est pas possible de base de stocker des vecteurs Numpy dans une base de données SQLite. Pour cela, nous allons créer un adaptateur qui va nous permettre de convertir nos vecteurs Numpy en un type que SQLite peut stocker. Pour cela, nous allons créer un fichier simple_vector_db/numpy_array_adapter.py et y créer une classe NumpyArrayAdapter qui va hériter de la classe types.TypeDecorator de SQLAlchemy.
# simple_vector_db/numpy_array_adapter.py
import numpy as np
from sqlalchemy import types
class NumpyArrayAdapter(types.TypeDecorator):
impl = types.LargeBinary
def process_bind_param(self, value, dialect):
if value is not None:
return value.tobytes()
def process_result_value(self, value, dialect):
if value is not None:
return np.frombuffer(value, dtype=np.float64)
Nous allons désormais pouvoir insérer des vecteurs dans notre base de données. Pour cela, nous allons ajouter une méthode insert à notre classe VectorDBSQLite. Cette méthode va prendre en entrée une liste de vecteurs et va les insérer dans la base de données.
# simple_vector_db/vector_db_sqlite.py
import numpy as np
class VectorDBSQLite():
...
def insert(self, vectors: list[np.ndarray], vector_ids: list[int] = None) -> None:
vector_objects = [Vector(data=array) for array in vectors]
self.session.add_all(vector_objects)
self.session.commit()
On peut créer un script main_sqlite.py pour tester une insertion. Nous allons créer une base de données SQLite dans un fichier vector_db.db et y insérer 1000 vecteurs de dimension (1, 3).
# main_sqlite.py
import numpy as np
from simple_vector_db.vector_db_sqlite import VectorDBSQLite
DB_FILENAME = "vector_db.db"
vector_db = VectorDBSQLite(db_filename=DB_FILENAME)
N_VECTORS = 1000
VECTORS_SHAPE = (1, 3)
def insert_vectors(n_vectors: int, vectors_shape: tuple[int, int]) -> None:
vectors_to_insert = [np.random.rand(*vectors_shape) for _ in range(n_vectors)]
vector_db.insert(vectors_to_insert)
if __name__ == "__main__":
insert_vectors(N_VECTORS, VECTORS_SHAPE)
Nous allons maintenant implémenter la méthode search de notre classe VectorDBSQLite. Cette méthode va prendre en entrée un vecteur de requête et un nombre k et va retourner les k vecteurs les plus similaires à la requête.
# simple_vector_db/vector_db_sqlite.py
from typing import List, Tuple
from simple_vector_db.distances import cosine_similarity
class VectorDBSQLite():
...
def search_without_index(
self, query_vector: np.ndarray, k: int
) -> List[Tuple[int, float]]:
vectors = self.session.query(Vector).all()
similarities = [
(vector.id, self.metric["metric"](query_vector, vector.data))
for vector in vectors
]
similarities.sort(key=lambda x: x[1], reverse=self.metric["reverse_sort"])
top_similarities = similarities[:k]
return top_similarities
De même que tout à l'heure avec l'implémentation en mémoire, il est possible de changer la distance utilisée pour la recherche. Ici, nous utilisons la similarité cosinus.
Nous pouvons retourner sur notre fichier main_sqlite.py et tester la recherche de vecteurs similaires. Pour cela, nous allons utiliser le décorateur timeit que nous avons créé plus tôt pour mesurer le temps d'exécution de la recherche, car nous comparerons cette recherche sans index avec une recherche avec index, que nous créerons par la suite.
Pour rendre l'exécution reproductible, nous supprimons le fichier vector_db.db à la fin de l'exécution du script. N'hésitez pas à ajouter une seed pour rendre l'exécution idempotente.
# main_sqlite.py
import logging
import shutil
import numpy as np
from simple_vector_db.vector_db_sqlite import VectorDBSQLite
from utils.flex_logging import stream_handler
from utils.timing import timeit
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
DB_FILENAME = "vector_db.db"
vector_db = VectorDBSQLite(db_filename=DB_FILENAME)
N_VECTORS = 1000
VECTORS_SHAPE = (1, 3)
QUERY_VECTOR = np.array([0.15, 0.25, 0.35])
K_SIMILAR_VECTORS = 5
def insert_vectors(n_vectors: int, vectors_shape: tuple[int, int]) -> None:
vectors_to_insert = [np.random.rand(*vectors_shape) for _ in range(n_vectors)]
vector_db.insert(vectors_to_insert)
@timeit
def perform_search_without_index():
similar_vectors = vector_db.search_without_index(QUERY_VECTOR, k=K_SIMILAR_VECTORS)
logger.info(f"Most {K_SIMILAR_VECTORS} Similar vectors: {similar_vectors}")
if __name__ == "__main__":
insert_vectors(N_VECTORS, VECTORS_SHAPE)
perform_search_without_index()
shutil.os.remove(DB_FILENAME)
Vous pouvez désormais lancer le script et voir le temps d'exécution de la recherche sans index. N'hésitez pas à changer les valeurs du vecteur de requête, les dimensions, le nombre de vecteurs similaires à retourner, etc.
Nous allons désormais créer un index pour accélérer la recherche des vecteurs les plus similaires.
Pour cela, nous allons ajouter une méthode create_kmeans_index à notre classe VectorDBSQLite. Cette méthode va créer des clusters avec la méthode Kmeans en se basant sur la colonne data de nos vecteurs.
# simple_vector_db/vector_db_sqlite.py
from sklearn.cluster import KMeans
class VectorDBSQLite():
...
def create_kmeans_index(self, n_clusters: int) -> np.ndarray:
vectors = self.session.query(Vector).all()
vector_arrays = [vector.data for vector in vectors]
kmeans = KMeans(n_clusters=n_clusters, random_state=0, n_init="auto")
kmeans.fit_predict(vector_arrays)
centroids = kmeans.cluster_centers_
self.insert_centroids(centroids)
self.insert_indexed_vectors(vector_arrays, kmeans.labels_)
return centroids
Il faut donc créer deux nouvelles méthodes insert_centroids et insert_indexed_vectors qui vont insérer les centroids et les vecteurs indexés dans la base de données.
Créons d'abord deux nouvelles classes Centroid et IndexedVector. Les centroïdes ont un id et des coordonnées ; les vecteurs indexés ont un id, des coordonnées et un cluster d'appartenance.
# simple_vector_db/vector_db_sqlite.py
class Centroid(Base):
__tablename__ = "centroids"
id = Column(Integer, primary_key=True)
data = Column(NumpyArrayAdapter)
def __init__(self, id, data):
self.id = id
self.data = data
class IndexedVector(Base):
__tablename__ = "indexed_vectors"
id = Column(Integer, primary_key=True)
data = Column(NumpyArrayAdapter)
cluster = Column(Integer)
def __init__(self, data, cluster):
self.data = data
self.cluster = cluster
Les méthodes insert_centroids et insert_indexed_vectors de la classe VectorDBSQLite sont assez similaires à la méthode insert que nous avons implémentée plus tôt.
# simple_vector_db/vector_db_sqlite.py
class VectorDBSQLite():
...
def insert_centroids(self, centroids) -> None:
centroid_objects = [
Centroid(id=id, data=centroid_coordinates)
for id, centroid_coordinates in enumerate(centroids) # because centroids id have to start at O
]
self.session.add_all(centroid_objects)
self.session.commit()
def insert_indexed_vectors(self, vectors, clusters) -> None:
indexed_vector_objects = [
IndexedVector(
data=vector_coordinates, cluster=int(cluster)
)
for vector_coordinates, cluster in zip(vectors, clusters)
]
self.session.add_all(indexed_vector_objects)
self.session.commit()
Nous allons désormais pouvoir implémenter la méthode search_in_kmeans_index de notre classe VectorDBSQLite. Cette méthode va prendre en entrée un vecteur de requête et un nombre k et va retourner les k vecteurs les plus similaires à la requête.
Il suffit alors de requêter la base de données pour récupérer les centroïdes, de trouver le centroïde le plus proche de la requête, de récupérer les vecteurs indexés appartenant à ce cluster et de calculer la similarité entre la requête et ces vecteurs.
# simple_vector_db/vector_db_sqlite.py
class VectorDBSQLite():
...
def search_in_kmeans_index(
self, query_vector: np.ndarray, k: int, n_probes=1
) -> Tuple[List[Tuple[int, float]], int]:
centroids = self.session.query(Centroid).all()
most_similar_centroid_id = self.find_most_similar_centroid(
query_vector, centroids, n_probes=n_probes
)
indexed_vectors = (
self.session.query(IndexedVector)
.filter(IndexedVector.cluster.in_(most_similar_centroid_id))
.all()
)
similarities = [
(indexed_vector.id, self.metric["metric"](query_vector, indexed_vector.data))
for indexed_vector in indexed_vectors
]
similarities.sort(key=lambda x: x[1], reverse=self.metric["reverse_sort"])
most_similar_vectors = similarities[:k]
return most_similar_vectors, most_similar_centroid_id
def find_most_similar_centroid(self, query_vector, centroids, n_probes=1) -> list:
centroid_similarities = [
(centroid.id, self.metric["metric"](query_vector, centroid.data))
for centroid in centroids
]
centroid_similarities.sort(key=lambda x: x[1], reverse=self.metric["reverse_sort"])
most_similar_centroid_ids = self.subset_most_similar_centroids(centroid_similarities,n_probes=n_probes)
return most_similar_centroid_ids
@staticmethod
def subset_most_similar_centroids(centroids_similarities, n_probes=1):
most_similar_centroid_ids = [tuple[0] for tuple in centroids_similarities[:n_probes]]
return most_similar_centroid_ids
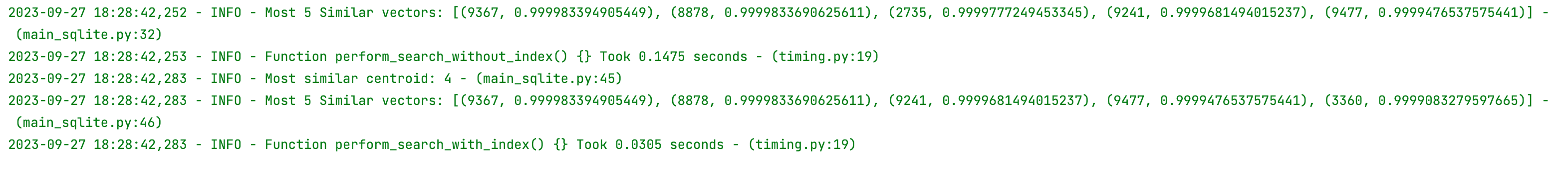
Retournons désormais dans notre fichier main_sqlite.py pour tester notre implémentation.
# main_sqlite.py
import logging
import shutil
import numpy as np
from simple_vector_db.vector_db_sqlite import VectorDBSQLite
from utils.flex_logging import stream_handler
from utils.timing import timeit
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
DB_FILENAME = "vector_db.db"
vector_db = VectorDBSQLite(db_filename=DB_FILENAME)
N_VECTORS = 1000
VECTORS_SHAPE = (1, 3)
QUERY_VECTOR = np.array([0.15, 0.25, 0.35]) # should be of same size as vectors shape
K_SIMILAR_VECTORS = 5
N_CLUSTERS = 5
def insert_vectors(n_vectors: int, vectors_shape: tuple[int, int]) -> None:
vectors_to_insert = [np.random.rand(*vectors_shape) for _ in range(n_vectors)]
vector_db.insert(vectors_to_insert)
@timeit
def perform_search_without_index():
similar_vectors = vector_db.search_without_index(QUERY_VECTOR, k=K_SIMILAR_VECTORS)
logger.info(f"Most {K_SIMILAR_VECTORS} Similar vectors: {similar_vectors}")
def create_index():
centroids = vector_db.create_kmeans_index(n_clusters=N_CLUSTERS)
logger.info(centroids)
@timeit
def perform_search_with_index():
most_similar_vectors, most_similar_centroid = vector_db.search_in_kmeans_index(
query_vector=QUERY_VECTOR, k=K_SIMILAR_VECTORS
)
logger.info(f"Most similar centroid: {most_similar_centroid}")
logger.info(f"Most {K_SIMILAR_VECTORS} Similar vectors: {most_similar_vectors}")
if __name__ == "__main__":
insert_vectors(N_VECTORS, VECTORS_SHAPE)
create_index()
perform_search_without_index()
perform_search_with_index()
shutil.os.remove(DB_FILENAME)
Félicitations, vous pouvez remarquer que la recherche avec index est beaucoup plus rapide que la recherche sans index !
Pour accélérer encore plus la recherche des vecteurs les plus similaires, nous allons implémenter la Product Quantization. Pour cela, nous allons créer un fichier simple_vector_db/vector_db_sqlite_pq.py et y créer une classe VectorDBSQLitePQ, qui héritera de VectorDBSQLite. Nous lui ajoutons un Vector Quantizer pour compresser les vecteurs, que nous allons implémenter par la suite :
# simple_vector_db/vector_db_sqlite_pq.py
from simple_vector_db.vector_db_sqlite import VectorDBSQLite
from simple_vector_db.quantization.vector_quantizer import VectorQuantizer
class VectorDBSQLitePQ(VectorDBSQLite):
def __init__(self, db_filename: str, m_chunks: int, nb_subspace_centroids: int):
super(VectorDBSQLitePQ, self).__init__(db_filename)
self.db_quantizer = VectorQuantizer(m_chunks, nb_subspace_centroids)
...
Nous allons désormais implémenter le Vector Quantizer. Pour cela, nous allons créer un fichier simple_vector_db/quantization/vector_quantizer.py et y créer une classe VectorQuantizer. Voici l'initialisation de la classe :
# simple_vector_db/quantization/vector_quantizer.py
import numpy as np
import logging
from utils.flex_logging import stream_handler
from sklearn.cluster import KMeans
from typing import Dict
from simple_vector_db.distances import euclidean_distance
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
class VectorQuantizer:
def __init__(self, m_chunks: int, nb_subspace_centroids: int):
self.m_chunks = m_chunks
self.nb_subspace_centroids = nb_subspace_centroids
self.codebook: Dict[int, dict[int, np.array]] = {}
...
Comme décrit dans notre article, les différentes étapes de quantization d'un vecteur sont les suivantes :
- Prendre un vecteur "v" de dimension "d". Diviser ce vecteur en "m" sections de taille égale, où "m" est généralement un nombre choisi à l'avance (plus m est grand et plus l'on compresse). Chaque section devient un sous-espace vectoriel de dimension "d/m". Par exemple, si "d" est égal à 32 et "m" est égal à 4, chaque sous-espace aura une dimension "d/m" de 8.
- Au sein de chaque chunk, effectuer une segmentation k-means en "k" clusters. Par exemple, si "k" est égal à 8 et "m" est égal à 4, on obtient un total de "k * m" clusters, soit 32 clusters au total, avec 8 clusters par sous-espace. Chaque cluster aura un centroïde comme dans la méthode précédente.
- Créer le codebook, qui sera essentiel pour décompresser les vecteurs ultérieurement. Le codebook contient les vecteurs représentant les centroïdes de chaque cluster. Il y aura "k*m", soit dans ce cas 32 vecteurs dans le codebook, un pour chaque cluster de chaque section du vecteur d'origine.
- Pour compresser le vecteur d'origine, attribuer le centroïde le plus proche dans chaque sous-espace de chaque section du vecteur d'origine. Ainsi, le vecteur "v" d'origine, qui avait "d" dimensions, est maintenant compressé en "m" composantes, chacune des dimensions prenant la valeur de l'ID unique du cluster (de 0 à "(k*m)-1").
# simple_vector_db/quantization/vector_quantizer.py
import numpy as np
import logging
from utils.flex_logging import stream_handler
from sklearn.cluster import KMeans
from typing import Dict
from simple_vector_db.distances import euclidean_distance
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
class VectorQuantizer:
def __init__(self, m_chunks: int, nb_subspace_centroids: int):
self.m_chunks = m_chunks
self.nb_subspace_centroids = nb_subspace_centroids
self.codebook: Dict[int, dict[int, np.array]] = {}
def split_vector_into_chunks(self, input_vector: np.array):
vector_dimension = input_vector.shape[0]
if vector_dimension % self.m_chunks != 0:
logger.error(f"The vector's dimension {vector_dimension} is not divisible by {self.m_chunks}")
chunk_dimension = int(vector_dimension / self.m_chunks)
chunks = [input_vector[ch * chunk_dimension:(ch + 1) * chunk_dimension] for ch in range(self.m_chunks)]
return chunks
def compute_clusters_on_subspace(self, subspace_vectors: list[np.array], subspace_index: int):
kmeans = KMeans(n_clusters=self.nb_subspace_centroids, random_state=0, n_init="auto")
kmeans.fit_predict(subspace_vectors)
centroids = kmeans.cluster_centers_
predicted_clusters = kmeans.labels_
subspace_codebook = {}
for i, el in enumerate(centroids):
subspace_codebook[i] = el
self.codebook[subspace_index] = subspace_codebook
return centroids, predicted_clusters
def quantize_vectors(self, input_vectors: list[np.array]) -> list[np.array]:
input_vectors_matrix = np.array(input_vectors)
vector_dimension = input_vectors[0].shape[0]
if vector_dimension % self.m_chunks != 0:
logger.error(f"The vector's dimension {vector_dimension} is not divisible by {self.m_chunks}")
chunks = np.split(input_vectors_matrix, self.m_chunks, axis=1)
quantized_vector = [self.compute_clusters_on_subspace(chunk, i)[1] for i, chunk in enumerate(chunks)]
return np.array(quantized_vector).T
def rebuild_vector(self, input_vector: np.array):
rebuilt_vector = np.array([])
for subspace_index, chunk_centroid_id in enumerate(input_vector):
rebuilt_chunk = self.codebook[subspace_index][chunk_centroid_id]
rebuilt_vector = np.append(rebuilt_vector, rebuilt_chunk)
return rebuilt_vector
def compute_assymetric_distance_matrix(self, query_vector: np.array) -> dict[int, dict]:
chunks = self.split_vector_into_chunks(query_vector)
distance_matrix = {subspace_index: self.distance_chunk_centroids(chunk, self.codebook[subspace_index]) for
subspace_index, chunk in enumerate(chunks)}
return distance_matrix
def compute_distances_for_all_vectors(self, distance_matrix: dict[int, dict], quantized_vectors: list[np.array]):
distance_list = []
for vector in quantized_vectors:
subspace_distances = np.array(
[distance_matrix[int(subspace_id)][int(cluster_id)] for subspace_id, cluster_id in enumerate(vector)]) # casting in int because of a bug in numpy with float64
distance_list.append(subspace_distances.sum())
return distance_list
@staticmethod
def distance_chunk_centroids(chunk: np.array, subspace_centroids: dict[int, np.array]) -> np.array:
return {centroid_id: euclidean_distance(chunk, subspace_centroids[centroid_id]) for centroid_id in
subspace_centroids.keys()}
Le Vector Quantizer est désormais implémenté et avant de l'ajouter dans notre vector db, nous pouvons déjà tester qu'il fonctionne seul. Nous pouvons l'appeler dans un script main_quantizer.py :
Ainsi ici, nous chargeons les données MNIST, nous quantisons les vecteurs, nous reconstruisons un vecteur à partir des vecteurs quantisés, nous calculons l'erreur de compression et nous recherchons les vecteurs les plus similaires à un vecteur de requête.
# main_quantizer.py
import logging
import numpy as np
from sklearn.datasets import load_digits
from simple_vector_db.quantization.vector_quantizer import VectorQuantizer
from utils.flex_logging import stream_handler
from sklearn.metrics import mean_squared_error
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
quantizer = VectorQuantizer(m_chunks=16, nb_subspace_centroids=32)
def perform_quantization():
vectors_to_quantize = load_digits().data # Données MNIST
labels = load_digits().target
idx_query_vector = 11
query_vector = vectors_to_quantize[idx_query_vector]
quantized_vectors = quantizer.quantize_vectors(vectors_to_quantize)
logger.info(f"quantized vector (centroids ids): {quantized_vectors[idx_query_vector]}")
codebook = quantizer.codebook
logger.info(f"Current Codebook: {codebook}")
logger.info(f"original vector: {query_vector}")
rebuilt_vector = quantizer.rebuild_vector(quantized_vectors[idx_query_vector])
logger.info(f"rebuilt vector: {rebuilt_vector}")
compression_mse = mean_squared_error(query_vector, rebuilt_vector)
logger.info(f"Mean squared error of compression for first vector {compression_mse}")
knn = find_knn_with_quantization(query_vector, quantizer, quantized_vectors)
logger.info("Knn results:"+str(knn))
logger.info(f'Label for the query vector is {labels[idx_query_vector]}')
nn_ids = [tuple[1] for tuple in knn]
nn_labels = labels[nn_ids]
logger.info(f'Labels of the 20 nearest neighbors are {nn_labels}')
def find_knn_with_quantization(query_vector: np.array, quantizer: VectorQuantizer,
quantized_vectors: list[np.array], k: int = 20):
distance_matrix = quantizer.compute_assymetric_distance_matrix(query_vector)
distances = quantizer.compute_distances_for_all_vectors(distance_matrix, quantized_vectors)
distances_ids = sorted(list(zip(distances, range(len(distances)))), key=lambda x: x[0])
distances_ids = distances_ids[0:k]
return distances_ids
if __name__ == "__main__":
perform_quantization()
L'exécution de ce script montre que l'implémentation du Vector Quantizer fonctionne bien.
Maintenant que nous avons implémenté le Vector Quantizer, nous pouvons compléter la classe VectorDBSQLitePQ. Nous allons ajouter les méthodes insert qui va insérer des vecteurs dans la base de données en les quantisant avec le Vector Quantizer, et search qui va rechercher les vecteurs les plus similaires en quantisant le vecteur de requête et en calculant la distance entre les vecteurs quantisés.
# simple_vector_db/vector_db_sqlite_pq.py
import numpy as np
from simple_vector_db.quantization.vector_quantizer import VectorQuantizer
from simple_vector_db.vector_db_sqlite import VectorDBSQLite, Vector
class VectorDBSQLitePQ(VectorDBSQLite):
def __init__(self, db_filename: str, m_chunks: int, nb_subspace_centroids: int):
super(VectorDBSQLitePQ, self).__init__(db_filename)
self.db_quantizer = VectorQuantizer(m_chunks, nb_subspace_centroids)
def insert(self, vectors: list[np.ndarray], vector_ids: list[int] = None):
quantized_vectors: list[np.ndarray] = self.db_quantizer.quantize_vectors(vectors)
super().insert(quantized_vectors, vector_ids)
def search_without_index(self, query_vector: np.ndarray, k: int):
quantized_vectors = self.session.query(Vector).all()
quantized_vectors_data = [vec.data for vec in quantized_vectors]
quantized_vectors_ids = [vec.id for vec in quantized_vectors]
distance_matrix = self.db_quantizer.compute_assymetric_distance_matrix(query_vector)
distances = self.db_quantizer.compute_distances_for_all_vectors(distance_matrix, quantized_vectors_data)
distances_ids = sorted(list(zip( quantized_vectors_ids,distances)), key=lambda x: x[1])
ann_results = distances_ids[0:k]
return ann_results
Nous pouvons créer un nouveau fichier main_sqlite_pq.py, dans lequel nous allons tester l'implémentation de la base de données vecteurs SQLite avec Product Quantization.
# main_sqlite_pq.py
import logging
import shutil
from sklearn.datasets import load_digits
from simple_vector_db.vector_db_sqlite_pq import VectorDBSQLitePQ
from utils.flex_logging import stream_handler
logger = logging.getLogger(__name__)
logger.addHandler(stream_handler)
logger.setLevel(logging.INFO)
DB_FILENAME = "vector_db.db"
IDX_QUERY_VECTOR = 11
K_SIMILAR_VECTORS = 10
M_CHUNKS = 16
N_CENTROIDS = 32
def perform_query_on_quantized_db():
vectordbPQ = VectorDBSQLitePQ(DB_FILENAME, m_chunks=M_CHUNKS, nb_subspace_centroids=N_CENTROIDS)
vectors_to_quantize = load_digits().data
vectordbPQ.insert(vectors_to_quantize, list(range(0, len(vectors_to_quantize))))
idx_query_vector = 11
query_vector = vectors_to_quantize[idx_query_vector]
results = vectordbPQ.search_without_index(query_vector, k=K_SIMILAR_VECTORS)
logger.info("Results Vector are:" + str(results))
if __name__ == "__main__":
perform_query_on_quantized_db()
shutil.os.remove(DB_FILENAME)
Et voici le run de ce fichier :
Bravo ! Si vous êtes arrivés jusqu'ici, vous avez implémenté une base de données vecteurs SQLite avec Product Quantization. Vous pouvez désormais l'utiliser pour accélérer vos recherches de vecteurs similaires. N'hésitez pas à relire l'ensemble des concepts présentés dans l'article.
Ce code n'a pas vocation à être utilisé en production, mais plutôt à vous donner une idée de comment fonctionne une base de données vecteurs.
Si vous souhaitez aller plus loin, vous pouvez implémenter d'autres méthodes de compression de vecteurs, comme la Multi-Index Hashing, ou encore la recherche de vecteurs similaires avec des arbres de recherche, comme les KD-Trees ou les Ball Trees.
De même, si vous souhaitez implémenter des méthodes d'évaluation, n'hésitez pas à regarder sur notre branche main pour voir l'implémentation que nous avons créée pour tous ces jolis schémas que vous pouvez voir dans notre article.